
Partie I :
Contexte
biologique
La bactérie la plus fréquemment retrouvée chez l’homme est le colibacille ou Escherichia coli. Cette bactérie est l’hôte normal de l’intestin. Elle peut être responsable d'infections des voies urinaires, de gastro-entérites ou d'infections nosocomiales. Escherichia coli est aussi la bactérie la mieux caractérisée, devenue pour les microbiologistes le modèle d’étude par excellence.
Escherichia coli peut être à son tour la cible d’agents nuisibles. Il peut s’agir d’antibiotiques bloquant son activité, de toxines protéiques (colicines) secrétées par des colonies concurrentes ou encore de virus parasitant sa machinerie cellulaire à des fins de reproduction (bactériophages).
Cinq ans après le séquençage du génome complet de la souche de référence[1] d’Escherichia coli (Blattner et al., 1997) et alors que la résistance des bactéries aux antibiotiques (Walsh, 2000) constitue un problème de plus en plus préoccupant, des zones d’ombres subsistent dans notre connaissance des mécanismes d’entrée de macromolécules à travers l’enveloppe d’Escherichia coli. Nous nous intéressons ici à l’internalisation d’une part des colicines et d’autre part de l’ADN de bactériophages filamenteux.
L’enveloppe de la bactérie représente une barrière contre l’internalisation des molécules de grande taille. Comment les agents infectieux parviennent-ils à franchir la membrane externe d’Escherichia coli depuis l’extérieur (translocation) ? Cette question nous conduit à examiner l’enveloppe des bactéries à Gram négatif dans un premier chapitre. Puis, nous présentons les colicines et les bactériophages avant de retracer les grands progrès de la connaissance concernant la translocation des colicines et de l’ADN des bactériophages. Nous terminons cette partie par un chapitre présentant de manière plus détaillée les deux grands systèmes de translocation.
L’enveloppe des bactéries à Gram négatif est constituée de deux membranes (interne et externe) délimitant l’espace périplasmique, dans lequel se trouve le peptidoglycane (figure 1).

Figure 1 : Structure
de l’enveloppe des bactéries à Gram négatif ;
figure adaptée de (Huijbregts et al., 2000)
La membrane interne de la bactérie délimite le cytoplasme. Encore appelée membrane cytoplasmique, cette bicouche phospholipidique est le siège du métabolisme respiratoire de la bactérie. Elle contient les protéines des chaînes de transfert d’électrons, qui provoquent un flux de protons conduisant à l’établissement d’une différence de potentiel électrochimique et de pH de part et d’autre de la membrane (force proton-motrice). La membrane interne contient également des perméases commandant l’import sélectif de molécules dans le cytoplasme. Il s’agit d’un transport actif, consommateur d’énergie.
La membrane interne des bactéries est recouverte d’une couche de muréine (ou peptidoglycane). Cette paroi est poreuse mais assure la rigidité de la bactérie et lui confère sa forme.
Parmi les bactéries, celles à Gram négatif possèdent une membrane lipidique supplémentaire, la membrane externe, dont la couche de lipides extérieure est composée de lipopolysaccharides (LPS). Les LPS porte l’antigène O qui définit les différents sérotypes bactériens.
La membrane externe délimite avec la membrane interne un espace appelé périplasme. L’épaisseur du périplasme est estimée entre 70 et 250 Å (Van Wielink et Duine, 1990). Le périplasme est un milieu oxydant[2] : la formation des ponts disulfures dans les protéines est assurée par l’enzyme DsbA, régénérée par la protéine membranaire DsbB couplée à la chaîne de transfert d’électrons de la membrane interne (Glockshuber, 1999).
La membrane externe joue un rôle de filtre pour la bactérie (Nikaido et Vaara, 1985). Des porines permettent la diffusion de petits solutés (ions, sucres de moins de 600 Da) entre le milieu extérieur et le périplasme, tout en protégeant la bactérie des principaux agents nuisibles (bactériophages, colicines, protéases) (Nikaido, 1993). La quasi totalité de ces porines ont une structure tridimensionnelle en tonneau ß (Koebnik et al., 2000), la sélectivité des solutés (taille et polarité ionique) pouvant être assurée par une zone de constriction à l’intérieur de chaque tonneau ß (Cowan et al., 1992).
Les porines les plus répandues chez E. coli sont trimériques comme OmpF (outer membrane protein F, figure 2), OmpC ou PhoE.

Figure 2 : Représentation
de la porine OmpF d’après modélisation
moléculaire (Watanabe et
al., 1997) à partir de la structure cristallographique (Cowan et al., 1995). (a) vue du
monomère depuis l’intérieur de la membrane (b) vue du
trimère le long de l’axe de symétrie d’ordre 3
représenté par un point ;
figure reproduite de (Watanabe et al., 1997).
D’autres porines de la membrane externe sont monomériques. C’est le cas de OmpA (Sugawara et Nikaido, 1992), qui jouerait également un rôle structural dans la stabilité membranaire en se liant au peptidoglycane (De Mot et Vanderleyden, 1994; Koebnik, 1995).
Certaines porines de la membrane externe disposent, en plus, d’un site de fixation spécifique à certains substrats. Il en résulte que la concentration locale de ces substrats est augmentée, ce qui accélère leur diffusion vers le périplasme. Il en est ainsi des maltodextrines (Boos et Shuman, 1998) à travers la maltoporine trimérique LamB (Schirmer et al., 1995), du sucrose à travers ScrY (Schulein et al., 1991), des nucléosides à travers Tsx (Maier et al., 1988). Il s’agit cependant d’un transport passif (ne nécessitant aucune source d’énergie).
Au contraire, certaines protéines membranaires sont des récepteurs spécifiques de haute affinité permettant l’import actif de substrats et leur concentration dans le périplasme. C’est le cas de BtuB (Reynolds et al., 1980) pour l’import de la cyano-cobalamine[3], de FepA (Buchanan et al., 1999), FhuA (Ferguson et al., 1998) et FecA (Ferguson et al., 2002) (figure3) pour l’import des sidérophores[4]. Puisque la membrane externe ne dispose d’aucune source d’énergie, le fonctionnement de ces récepteurs nécessite un couplage énergétique avec la membrane interne, réalisé par l’intermédiaire du complexe protéique TonB-ExbBD.

Figure 3 : Représentation du récepteur FecA en présence d’un ferrichrome, d’après la structure cristallographique. Figure reproduite de (Ferguson et al., 2002) (a) vue depuis l’intérieur de la membrane, les résidus 621 à 723 ayant été retirés pour faire apparaître l’intérieur de la porine ; (b) vue le long de l’axe.
Enfin, d’autres protéines intégrales de la membrane externe participent à l’export moléculaire comme OMPLA[5] (Snijder et al., 1999) ou comme TolC (Koronakis et al., 2000).

Figure 4 : Structure
de protéines de la membrane externe ;
figure reproduite de (Andersen et al., 2000).
Des lipoprotéines sont également ancrées dans la membrane externe : la lipoprotéine majeure Lpp est aussi liée de manière covalente au peptidoglycane, participant à la stabilisation de l’enveloppe bactérienne. Cette protéine est l’une des plus abondantes dans la bactérie (environ 7,2 x 105 exemplaires par cellule) et son altération provoque une hypersensibilité aux détergents, la formation de vésicules et le largage de protéines périplasmiques dans le milieu externe (Yem et Wu, 1978). La protéine Lpp mature forme un trimère hélicoïdal (Shu et al., 2000).
Escherichia coli est la cible d’agents nuisibles qui, en franchissant le membrane externe ou l’enveloppe bactérienne, peuvent provoquer la mort (agents bactéricides) ou seulement l’arrêt de la croissance (agents bactériostatiques) de la bactérie.
Certains antibiotiques inhibent la formation du peptidoglycane en occupant le site actif des enzymes assurant sa construction (cas des céphalosporines et des ß-lactames comme la pénicilline ou l’ampicilline) ou en se fixant au substrat peptidique (cas de la vancomycine). D’autres antibiotiques bloquent une des étapes de la synthèse des protéines (cas des tétracyclines et des aminoglycosides comme la kanamycine). D’autres composés s’attaquent aux enzymes responsables de la réplication et de la réparation de l’ADN (cas des fluoroquinolones) (Walsh, 2000).
Nous nous intéressons ici à l’entrée de deux autres types de macromolécules : d’une part, l’ADN des bactériophages filamenteux, s’introduisant dans E. coli et détournant sa machinerie cellulaire à des fins de reproduction ; d’autre part, les colicines, des toxines protéiques secrétées par des colonies concurrentes d’E. coli.
Les bactériophages filamenteux constituent une famille de phages à ADN simple-brin qui utilisent les bactéries à Gram négatif pour proliférer. Un tel phage est constitué d’une molécule d’ADN circulaire simple-brin, support d’une dizaine de gènes et enrobée d’une enveloppe protéique. Cette enveloppe (ou capside) de 800 à 2000 nm de long et de 6 nm de diamètre est constituée de plusieurs milliers de protéines alpha-hélicoïdales identiques (dites majeures) et de protéines mineures couvrant les extrémités de ce tube. Lors de l’infection, l’ADN du phage pénètre dans le cytoplasme de la bactérie où il est répliqué en ADN double-brin par la machinerie de l’hôte. L’ADN est alors dupliqué circulairement, les protéines d’enveloppe sont synthétisées et le tout est assemblé en complexe nucléo-protéique avant d’être extrudé à travers la membrane de la bactérie (Marvin, 1998).
La possibilité d’insérer dans l’ADN de phage une séquence d’ADN étrangère a ouvert la voie à d’importantes applications biotechnologiques. L’insertion d’un gène dans une région intergénique du phage permet de l’utiliser comme vecteur de clonage. La fusion en phase d’un ADN étranger avec un gène codant pour une protéine mineure de capside permet de présenter une protéine étrangère à la surface du virus. De telles protéines peuvent alors être sélectionnées dans une bibliothèque sur la base de leurs propriétés d’interaction avec une molécule cible immobilisée (phage display, figure 5). Il est également possible, en dissociant une protéine d’enveloppe en deux hybrides, de corréler le caractère infectieux du phage avec l’affinité entre les deux hybrides (Selectively Infective Phages, figure 5).
La protéine mineure de capside la plus utilisée pour ces applications est celle codée par le gène 3 et dénommée g3p. Elle est composée de trois domaines (N1 : 67 résidus, N2 : 131 résidus et CT : 150 résidus) espacés par des séparateurs de 19 et 39 résidus respectivement, riches en glycine. Le domaine CT ancre la protéine à l’extrémité du phage.

Figure 5 : Utilisation des bactériophages filamenteux pour l’identification de couples protéine-ligand ; figure reproduite de (Lubkowski et al., 1998).
La structure cristallographique des domaines N1 et N2 révèle qu’ils sont associés par l’intermédiaire d’un feuillet ß commun ((Lubkowski et al., 1998), figure 6).

Figure 6 : Structure
cristallographique des domaines N1 et N2 de la protéine g3p (Lubkowski et al., 1998) ;
figure tirée de la base de données PDB (code d’accession
1G3P)
Dans le groupe de phages Ff[6], la protéine g3p est responsable de l’import de l’ADN du phage par la bactérie. La liaison du domaine N2 de g3p au F–pilus d’Escherichia coli constitue la première étape de l’infection de la bactérie par le phage (étape de réception). Cette liaison a deux conséquences. D’une part, elle provoque, par un mécanisme inconnu, la rétraction du pilus, rapprochant ainsi de l’enveloppe bactérienne la protéine g3p. D’autre part, elle dégage le domaine N1 de g3p. Ce domaine assure la deuxième étape de l’infection : la pénétration de l’ADN à travers la membrane. Cette étape de translocation, aboutissant à l’entrée de l’ADN viral dans le cytoplasme bactérien, reste mal comprise.
Parmi les toxines anti-bactériennes, on appelle colicines les toxines protéiques produites par Escherichia coli sans modification post-traductionnelle, hormis la coupure du résidu de méthionine N-terminal. Cette définition ne recouvre pas les antibiotiques, qui sont des petits composés non-protéiques, ni les microcines, polypeptides qui subissent de nombreuses modifications post-traductionnelles. On dénombre actuellement 19 colicines[7] appelées colicine A[8], B, D, E1 à E9, Ia, Ib, K, M, N, colicine 5 et colicine 10, ces deux dernières ayant été découvertes plus récemment (Bradley et Howard, 1992; Pilsl et Braun, 1995). Leur masse moléculaire varie entre 29 et 75 kDa. (Braun et al., 1994)
Les colicines ont un effet létal sur Escherichia coli et certaines espèces bactériennes apparentées (par exemple Shigella et Salmonella). Leur fonction serait de défendre une niche écologique contre l’invasion de colonies concurrentes. La colonie qui produit une certaine colicine est protégée contre l’effet de cette colicine par la production conjointe d’une protéine antagoniste dite d’immunité. La colicine et la protéine d’immunité correspondante sont codées sur un même plasmide. La présence du plasmide distingue parmi les cellules bactériennes celles qui sont immunisées contre cette colicine.
Les colicines ne possèdent pas de séquence d’adressage et leur transport à travers l’enveloppe jusqu’au milieu extérieur ne passe pas par la machinerie de sécrétion classique[9] (Thanassi et Hultgren, 2000). En revanche, on trouve sur certains plasmides colicinogènes, à proximité du gène codant pour la colicine, un gène codant pour une protéine de lyse (d’une trentaine de résidus). La synthèse de cette lipoprotéine entraîne une perméabilisation de l’enveloppe bactérienne qui libère les colicines de façon non-spécifique.
Les colicines libérées dans le milieu peuvent tuer les bactéries concurrentes suivant différents mécanismes résumés dans le tableau ci-après. Les colicines A, B, E1, Ia, Ib, K, N, Col5 et Col10 forment des pores dans la membrane cytoplasmique de la bactérie, détruisant ainsi le potentiel de membrane indispensable à sa survie. Les colicines E2, E7, E8 et E9 dégradent l’ADN chromosomique de la bactérie (endodésoxyribonucléases[10]). Les colicines E3, E4, E6 et la cloacine DF13 coupent l’ARN ribosomal 16S, empêchant ainsi la biosynthèse des protéines. Les colicines D et E5 inhibent également la biosynthèse des protéines, mais par coupure de certains ARN de transfert (Ogawa et al., 1999). La colicine M inhibe la synthèse du peptidoglycane et de l’antigène O, entraînant la lyse cellulaire (Harkness et Olschlager, 1991).
|
Colicine |
Action létale |
|
A, B, E1, Ia, Ib, K, N, Col5, Col10 |
Déplétion du potentiel de membrane |
|
E2, E7, E8, E9 |
Dégradation d’ADN chromosomique |
|
E3, E4, E6, DF13 |
Coupure d’ARNr 16S |
|
D, E5 |
Coupure d’ARNt |
|
M |
Lyse cellulaire |
Tableau 1 : Action létale des différentes colicines.
L’infection de la bactérie par une colicine est un processus en trois étapes. L’action létale est précédée de la liaison de la colicine à un récepteur de la membrane externe puis de la traversée de l’enveloppe bactérienne (translocation). Le tableau suivant présente les récepteurs parasités par les différentes colicines lors de la première étape. L’étape de translocation est, elle, encore mal comprise, comme dans le cas des bactériophages.
|
Colicine |
Récepteur |
Fonction du récepteur |
|
A, E1 à E9 |
Récepteur de la vitamine B12 |
|
|
B, D |
Transport du fer |
|
|
M |
Transport du fer |
|
|
DF13 |
IutA |
Transport du fer |
|
Ia, Ib |
Cir |
Transport du fer (postulé) |
|
N |
Porine |
|
|
K, Col5, Col10 |
Canal à [désoxy]nucléosides |
Tableau 2 : Récepteurs d’E. coli parasités par les différentes colicines.
La structure primaire des colicines reflète leur mode d’action. La séquence protéique est divisée en trois domaines correspondant à chacune des trois étapes de l’infection. Le domaine central est responsable de la liaison au récepteur. Le domaine N-terminal est impliqué dans l’étape de translocation. Le domaine C-terminal porte l’activité létale (Baty et al., 1988). L’interaction de ce domaine avec la protéine d’immunité correspondante inhibe l’activité létale de la colicine.
La structure
tridimensionnelle de la colicine Ia a été résolue par
radiocristallographie (Wiener et al., 1997). Elle révèle trois domaines
fonctionnels séparés par deux longues hélices alpha de 160
Å chacune (figure 7a).

Figure 7 : (a) Structure tridimensionnelle de la colicine Ia entière à une résolution de 3.00 Å. (b) Structure tridimensionnelle de la colicine E3 en complexe avec sa protéine d’immunité à une résolution de 3.02 Å. (c) Structure tridimensionnelle du domaine de fixation au récepteur et du domaine létal la colicine N à une résolution de 3.10 Å. T = domaine de translocation, R = domaine de fixation au récepteur, L = domaine létal, I = protéine d’immunité. Figure construite à partir des fichiers de coordonnées respectifs 1CII, 1JCH et 1A87 de la base de données PDB.
La structure cristallographique de la colicine E3 a été résolue en complexe avec sa protéine d’immunité (Soelaiman et al., 2001). Elle présente une topologie analogue à celle de la colicine Ia : les trois domaines fonctionnels sont séparés par deux hélices alpha de 100 Å chacune (figure 7b).
La structure cristallographique de la colicine N entière a été déterminée (Vetter et al., 1998), mais son domaine de translocation n’apparaît pas, conformément aux données de dichroïsme circulaire et de fluorescence indiquant une absence de structure secondaire dans ce domaine (figure 7c).
D’autres structures tridimensionnelles de colicines ont été établies, en se limitant au domaine létal, soit seul (colicine A : (Parker et al., 1992), figure 8) soit en complexe avec une protéine d’immunité (colicine E7 avec Im7 : (Ko et al., 1999); colicine E9 avec Im9 : (Kuhlmann et al., 2000))

Figure 8 : Structure tridimensionnelle du domaine létal de la colicine A à une résolution de 2.4 Å d’après (Parker et al., 1992). Figure construite à partir du fichier de coordonnées 1COL de la base de données PDB.
Dans
le processus d’infection d’Escherichia coli par les
bactériophages comme par les colicines, c’est l’étape
de translocation qui reste encore la plus mal comprise.
Les colicines, par le mystère de leur translocation, se sont révélées être des outils d’investigation opportuns de l’enveloppe d’Escherichia coli.
Cette approche génétique (Nagel de Zwaig et Luria, 1967) a consisté à sélectionner des mutants d’Escherichia coli insensibles à l’action de colicines et à relier ces anomalies fonctionnelles à l’altération de gènes spécifiques. Certains mutants ont des défauts dans les récepteurs de la membrane externe, empêchant les colicines de se fixer à la bactérie. On parle, dans ce cas, de mutants “ résistants ” aux colicines. Chez d’autres mutants, les colicines sont adsorbées correctement, mais ne parviennent pourtant pas à inhiber la bactérie. Ces mutants sont dits “ tolérants ” aux colicines. Cette tolérance est indépendante du récepteur utilisé et de la cible sur laquelle la colicine exerce son action létale. Cela met en évidence l’existence d’un (ou plusieurs) système(s) protéique(s) intermédiaire(s) assurant la translocation des colicines adsorbées vers leur cible.
Une étude plus systématique de la tolérance aux colicines a permis une classification selon deux groupes mutuellement exclusifs (Davies et Reeves, 1975b), (Davies et Reeves, 1975a). Les colicines du groupe A (colicines A, E1 à E9, K, N, DF13) ainsi que les phages filamenteux f1, fd et M13 utilisent pour leur translocation les produits des gènes tol[11]. Les colicines du groupe B (colicines B, D, Ia, Ib, M, 5 et 10) et les phages T1[12] et F80 utilisent les protéines du système TonB-ExbBD (Lazdunski et al., 1998).
L’analyse des mutations responsables de la tolérance aux colicines du groupe A met en évidence 6 gènes tol parmi lesquels les gènes tolQ, tolR et tolA semblent jouer un rôle central. Les gènes tolB, tolC et tolZ semblent jouer des rôles annexes[13]. Des mutations dans le gène ompF sont également responsables de la tolérance à certaines colicines du groupe A. Par ailleurs, la translocation de l’ADN des phages filamenteux Ff dans le cytoplasme d’Escherichia coli nécessite également les produits des gènes tolQ, tolR et tolA (Webster, 1991).
Les gènes tolQ, tolR, tolA, tolB d’Escherichia coli sont organisés en un opéron contenant également les gènes pal, ybgC et ybgF (Vianney et al., 1996). Les avancées majeures dans le séquençage des génomes permettent d’observer que cet opéron est conservé chez bon nombre de bactéries à Gram négatif (figure 9,(Sturgis, 2001)).
Chez Vibrio cholerae, les produits des gènes tolQRA sont responsables de l’import de la toxine du choléra, véhiculée par le phage CTXf (Heilpern et Waldor, 2000).
Chez Pseudomonas aeruginosa, le gène tolA est l’un des 34 activés[14] lors de la formation des biofilms[15], qui rendent le pathogène résistant au traitement antimicrobien (Whiteley et al., 2001). Le produit de ce gène diminue l’affinité de la membrane externe pour les aminoglycosides en affectant la structure des lipopolysaccharides.
Dans l’opéron tol-pal, le gène tolA présente la plus grande variabilité d’une espèce à l’autre (Sturgis, 2001).

Figure 9 : Organisation des opérons tol-pal. Figure reproduite de (Sturgis, 2001)
En conclusion, l’étude de la tolérance aux colicines a permis de découvrir les gènes tol–pal, dont la fonction biologique reste encore obscure mais pour lesquels l’intérêt se confirme de jour en jour. La caractérisation des produits de ces gènes se poursuit donc activement.
Avant de présenter l’état des connaissances sur le système protéique Tol-Pal, nous commencerons par examiner le système TonB-ExbBD, dont la fonction est mieux caractérisée et avec lequel le système Tol-Pal présente de nombreux éléments de similitude.
Les colicines et les bactériophages utilisent le système TonB-ExbBD et le système Tol-Pal pour franchir la membrane externe d’Escherichia coli. La fonction du système TonB-ExbBD (transduction d’énergie pour l’import actif de substrats dans le périplasme) est bien identifiée. En revanche, celle du système Tol-Pal est encore imprécise. Ces deux systèmes présentent certains éléments de similitude.
L’import de certains substrats (cobalamine, sidérophores) dans le périplasme d’Escherichia coli se fait par l’intermédiaire de récepteurs de haute affinité situés dans la membrane externe. Un apport d’énergie est nécessaire pour dissocier le substrat du récepteur et le libérer dans le périplasme. Le système TonB-ExbBD assure la transduction d’énergie entre la membrane cytoplasmique de la bactérie et les récepteurs spécifiques de la membrane externe (Moeck et Coulton, 1998).
Le système TonB-ExbBD est constitué des protéines TonB[16] (239 résidus), ExbB (244 résidus) et ExbD (141 résidus). Les protéines ExbB et ExbD possèdent respectivement 3 et 1 segment(s) transmembranaire(s) et forment un complexe hétérohexamérique tête-bêche dans la membrane cytoplasmique (Higgs et al., 1998). L’extrémité N-terminale de la protéine TonB est ancré dans la membrane cytoplasmique et interagit avec ExbB et ExbD. Le reste de la protéine TonB s’étend à travers le périplasme jusqu’aux récepteurs de la membrane externe avec lesquels elle interagit (Kadner, 1990). Une quantité importante[17] de résidus de proline rigidifie la protéine (Evans et al., 1986), (Brewer et al., 1990). La structure cristallographique du domaine C-terminal de TonB révèle un dimère intimement entrelacé (figure 10,(Chang et al., 2001)) malgré des expériences de sédimentation suggérant que ce domaine se présente essentiellement sous forme monomérique en solution (Moeck et Letellier, 2001).

Figure 10 : Structure cristallographique du dimère du domaine C-terminal de la protéine TonB (résidus 165 à 237) déterminée par (Chang et al., 2001). Figure construite à partir du fichier de coordonnées 1IHR de la base de données PDB.
Le système TonB-ExbBD est responsable de l’import actif des sidérophores et de la cobalamine. Dans le paradigme actuel, le mécanisme de transport par transduction d’énergie peut être décrit de la manière suivante (figure 11).
Sous l’emprise de la force proton-motrice de la membrane interne, le complexe ExbB-ExbD maintient TonB dans une conformation “ active ” (Larsen et al., 1999). Au niveau de la membrane externe, un ligand vient se fixer dans un récepteur disponible et entraîne chez ce dernier des changements de conformations provoquant la fermeture du récepteur au milieu externe (Postle, 2002) et la présentation dans le périplasme d’un site de liaison à la protéine TonB dénommé ‘boîte TonB’ (Coggshall et al., 2001). La liaison de TonB actif au récepteur entraîne la libération du substrat dans le périplasme, le retour du récepteur à la conformation initiale et le retour de TonB “ passif ” en position initiale dans le complexe membranaire ExbB-ExbD. Un nouveau cycle peut alors commencer (Letain et Postle, 1997).

Figure 11 : Modèle de transduction d’énergie par l’intermédiaire de TonB (voir le texte). Figure reproduite de (Moeck et Coulton, 1998).
Les protéines TonB, ExbB et ExbD sont nécessaires à la translocation des colicines du groupe B et de l’ADN des phages T1 et F80 (Davies et Reeves, 1975a). Les colicines du groupe B contiennent dans leur domaine N-terminal une séquence de reconnaissance de TonB (‘boîte TonB’) semblable à celle des récepteurs de la membrane externe. Le traitement de bactéries par un peptide synthétique comportant cette séquence inhibe la croissance en milieu carencé en fer mais inhibe également l’infection par le phage F80 et les colicines B et Ia (Tuckman et Osburne, 1992). La liaison avec TonB par l’intermédiaire de cette séquence semble donc être une étape clé de la translocation des colicines du groupe B.
L’opéron tol-pal d’Escherichia coli (Vianney et al., 1996) code pour les protéines YbgC (134 résidus), TolQ (230 résidus), TolR (142 résidus), TolA (421 résidus), TolB (430 résidus), Pal (173 résidus) et YbgF (263 résidus).
YbgC est une protéine cytoplasmique. TolQ, TolR et TolA sont localisées dans la membrane interne. TolB et YbgF sont des protéines périplasmiques. Pal[18] est une lipoprotéine ancrée dans la membrane externe et associée de manière non covalente au peptidoglycane.
Les protéines TolQ, TolR et TolA forment un complexe au niveau de la membrane interne en interagissant par leurs segments transmembranaires (Journet et al., 1999). TolQ et TolR possèdent respectivement 3 et 1 segment(s) transmembranaire(s). TolA est divisée en trois domaines : le domaine N-terminal (TolA-I : résidus 1 à 47) est ancré dans la membrane cytoplasmique ; il est relié au domaine C-terminal (TolA-III : résidus 311 à 421) par l’intermédiaire d’une hélice alpha (TolA-II : résidus 48 à 310) (Levengood et al., 1991), (Derouiche et al., 1999).
Les protéines Pal et TolB forment un complexe associé à la membrane externe (Bouveret et al., 1995). Pal interagit avec TolB et OmpA. TolB interagit avec Pal, Lpp, OmpA[19] et avec les porines OmpF, OmpC, PhoE, LamB (Rigal et al., 1997).
Un lien a été établi entre les deux complexes du système Tol-Pal. Il a été montré que TolA-III (résidus 325 à 421) se lie à Pal (Cascales et al., 2000), que TolA-III (résidus 301 à 421) se lie à TolB (résidus 1 à 144) (Walburger et al., 2002), et que des mutations dans TolAIII (résidus F352I, F352C ou Y353I) pouvait être complémentées par une mutation[20] dans TolB (Dubuisson et al., 2002).
Par ailleurs, TolA-II (résidus 69 à 289) est nécessaire pour la liaison de TolA aux porines OmpF, PhoE et LamB (Derouiche et al., 1996). Enfin, TolA-II-III (résidus 66 à 421) se lie à YbgF (Walburger et al., 2002).

Figure 12 : Structure du système Tol-Pal d’Escherichia coli.
Des cristaux de la
protéine TolR ont pu être obtenus et la production de
dérivés substitués par la
séléno-méthionine devrait conduire à la
résolution de sa structure (Abergel et al., 2001). Les structures cristallographiques de Pal
et de TolB ont été déterminées (figure 13). TolB
est divisé en deux domaines : le domaine N-terminal (TolB-D1) et le
domaine C-terminal (TolB-D2) en forme de propulseur ((Abergel et al., 1999), (Carr et al., 2000)). La structure cristallographique
d’une protéine de fusion contenant le domaine N1 de g3p
et le domaine TolA-III a été résolue (figure 16,(Lubkowski et al., 1999)), ainsi que celle du domaine homologue TolA-III de Pseudomonas aeruginosa, qui présente seulement 20% de similarité de
séquence avec celui d’Escherichia coli (Witty et al., 2002).

Figure
13 : (a) Structure cristallographique d’un dimère de
Pal.
Figure reproduite de (Walburger, 2002)
(b) Structure cristallographique de TolB d’après (Abergel et al., 1999).
Figures construites à partir du fichier de coordonnées 1CRZ de la base de données PDB.
Le système Tol est nécessaire à la translocation des colicines du groupe A à travers le périplasme.
|
Colicine |
Système de translocation |
|
K |
OmpAF TolABQR |
|
E2, E3 |
BtuB OmpF TolABQR |
|
A, E6, E7, E8, E9 |
OmpF TolABQR |
|
N |
OmpF TolAQR |
|
E1 |
TolC TolAQ |
|
DF13 |
TolAQR |
Tableau 3 : Protéines impliquées dans la translocation des colicines du groupe A d’après (Lazdunski et al., 1998), (Bénédetti et al., 1989), (Gokce et al., 2000).
La translocation des colicines A et E1 est corrélée avec leur liaison à TolA-III[21], le domaine C-terminal de TolA. Cette liaison se fait par le domaine de translocation de chacune des colicines, avec des constantes de dissociation de 0,2 µM pour AT1, le domaine N-terminal de la colicine A et de 0,6 µM pour HS, le domaine N-terminal de la colicine E1 (Derouiche et al., 1997). Les résidus de la colicine A impliqués dans la liaison avec TolA sont compris entre 52 et 97 ; ceux impliqués dans l’interaction avec TolB sont compris entre 7 et 20 ; ceux impliqués dans l’interaction avec TolRII sont compris entre 7 et 14 (Journet et al., 2001).
Le modèle suivant a été proposé dans le cas de la colicine A (figure 14, (Lazdunski et al., 1998)). La liaison du domaine central de la colicine A avec le récepteur de la membrane externe entraîne le dépliement de la colicine (Duché et al., 1994). Le domaine N-terminal de la colicine A traverse le pore OmpF (Jeanteur et al., 1994) et interagit avec TolB et TolA-III (figure 14b). Le domaine C-terminal de la colicine A se retrouve alors dans le périplasme, par un mécanisme qu’il reste à élucider (figure 14c). Enfin, le domaine C-terminal de la colicine A s’insère dans la membrane cytoplasmique (figure 14d) et y forme un pore, tandis que le domaine central reste fixé à l’extérieur de la bactérie (Bénédetti et al., 1992).

Figure 14 : Modèle pour l’infection d’Escherichia coli par la colicine A. PG = peptidoglycane, T = domaine de translocation, R = domaine de fixation au récepteur, L = domaine létal. Figure adaptée de (Lazdunski et al., 1998).
Les protéines TolQ, TolR et TolA d’Escherichia coli sont nécessaires à la translocation de l’ADN des phages filamenteux Ff (Webster, 1991). Dans ce groupe de phages, la protéine d’enveloppe g3p est responsable de l’import de l’ADN viral par la bactérie. Après la liaison du domaine N2 de g3p au F-pilus d’Escherichia coli, le domaine N1 de g3p, libéré, peut se lier au domaine TolA-III (résidus 295 à 421) qui agit comme co–récepteur lors de l’infection (Riechmann et Holliger, 1997).

Figure 15 : Modèle pour les étapes initiales de l’infection d’Escherichia coli par les bactériophages du groupe Ff. Figure reproduite de (Lubkowski et al., 1999).
La structure cristallographique du complexe ainsi obtenu (figure 16) a été résolue grâce à une protéine de fusion contenant le domaine N1 de g3p et le domaine TolA-III (Lubkowski et al., 1999). Néanmoins, le mécanisme de translocation reste inexpliqué.
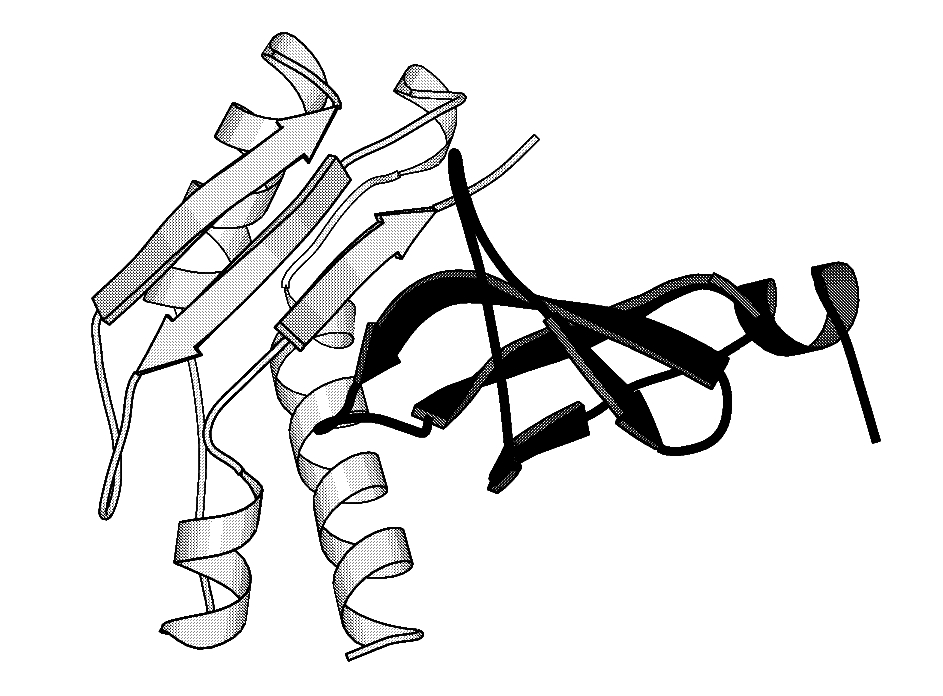
Figure 16 : Structure cristallographique d’une protéine de fusion contenant le domaine N1 de g3p (noir) et le domaine TolA-III (blanc) d’après (Lubkowski et al., 1999). Figure construite à partir du fichier de coordonnées 1TOL de la base de données PDB.
La fonction du système Tol-Pal (Lloubès et al., 2001) est encore incertaine. Une fonction liée à la biogenèse des composants de la membrane externe a été suggéré par l’interaction de TolA avec certaines porines (Derouiche et al., 1996) et par son rôle dans l’expression de l’antigène O (Gaspar et al., 2000). Par ailleurs, le système Tol-Pal joue un rôle capital dans le maintien de l’intégrité de la membrane externe. En effet, on constate chez des mutants tol et pal une hypersensibilité de la bactérie aux détergents et aux antibiotiques (Davies et Reeves, 1975a) ainsi que le déversement dans le milieu extérieur de protéines périplasmiques (Lazzaroni et al., 1999) et de vésicules (figure 17, (Bernadac et al., 1998)). Il est à noter que ce phénotype multiple n’a pas d’équivalent dans le système TonB.

Figure 17 : Section ultrafine d’un mutant tolA mettant en évidence la formation de vésicules. Figure reproduite de (Bernadac et al., 1998)
Le rôle du domaine TolA-III (résidus 291 à 421) dans l’intégrité de la membrane externe a été mis en évidence par (Levengood et al., 1993). La présence de ce domaine soluble en excès dans le périplasme provoque une hypersensibilité au deoxycholate, la fuite de protéines périplasmiques et augmente la tolérance aux colicines du groupe A. Il a été suggéré que le rôle de TolA soit de rapprocher les deux membranes, facilitant le transport de composés vers la membrane externe. Cette hypothèse est renforcée par la localisation préférentielle des protéines Tol aux sites de contact de l’enveloppe bactérienne (Guihard et al., 1994).
Outre leur rôle-clé dans la translocation des colicines et de l’ADN de bactériophages, le système Tol et le système TonB-ExbBD présentent plusieurs éléments de similitude.
Tout d’abord, la topologie des protéines TolA, TolQ et TolR est identique à celle des protéines TonB, ExbB et ExbD respectivement, TolA et TonB étant ancrées dans la membrane interne et traversant le périplasme.
En outre, TolQ et TolR présentent de fortes similarités de séquence avec ExbB et ExbD respectivement (Eick-Helmerich et Braun, 1989). La sensibilité aux colicines est conservée par complémentation croisée entre TolQ-TolR et ExbB-ExbD : un système ExbB-ExbD-TolA permet l’infection par les colicines du groupe A et un système TolQ-TolR-TonB permet l’infection par les colicines du groupe B (Braun et Herrmann, 1993).
Par ailleurs, la force proton-motrice, par l’intermédiaire de ExbB et ExbD, maintient TonB dans une conformation permettant l’interaction avec un récepteur de la membrane externe. De manière comparable, il a été découvert récemment que certains changements de conformation de TolA (Germon et al., 2001) ainsi que l’interaction entre TolA et Pal in vivo (Cascales et al., 2000), (Cascales et al., 2001) dépendaient de la force proton-motrice et des protéines TolQ et TolR.
Plus récemment encore, l’étude cristallographique de la structure du domaine III de TolA de Pseudomonas aeruginosa a mis en lumière une analogie structurale frappante entre ce domaine, le domaine correspondant d’E. coli et un pseudo-protomère de TonB d’E. coli (Witty et al., 2002).